KAIST-서울대, 새로운 맞춤형 대장암 치료법 제시
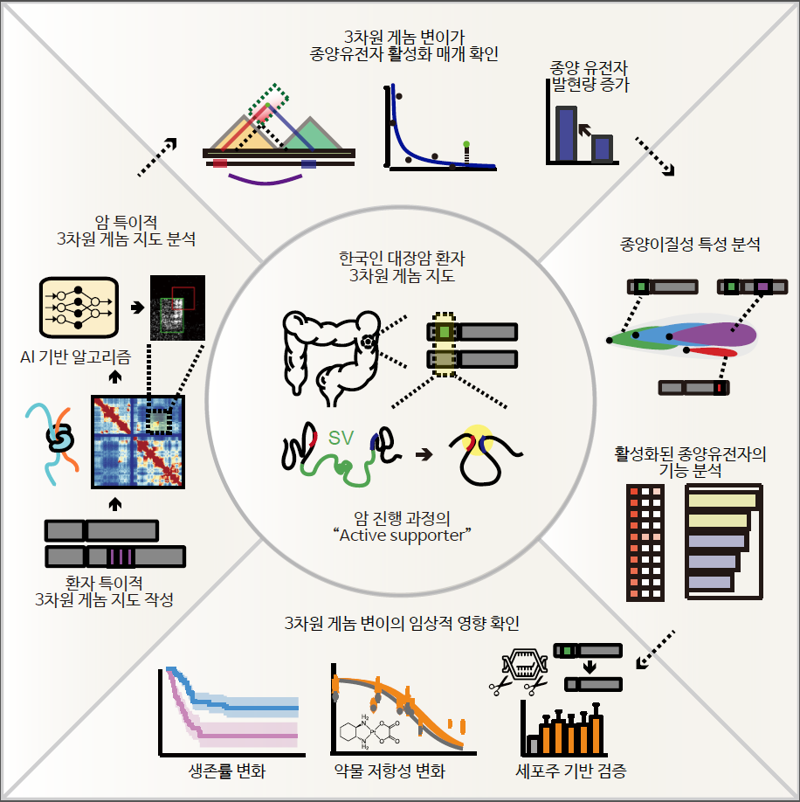
정인경 KAIST 교수는 24일 "기존의 점돌연변이나 유전체 변이만으로는 설명이 어려운 암 유전체를 3차원 게놈 구조 관점에서 재해독하고 신규 암 타겟을 발굴할 수 있는 수 있는 새로운 접근법을 제시했다"고 설명했다.
연구진은 이 게놈 지도로 암 세포 특이적인 유전자 조절 현상을 통해 특정 종양유전자들이 과하게 표출되는 현상을 밝혀냈다. 또한 김태유 서울대 교수는 "이 결과는 개별 암 환자들마다 서로 다르게 나타나는 종양 이질성을 이해하는 데 매우 중요한 요소가 될 수 있으며, 이를 이용한 환자 맞춤형 치료 연구의 시발점이 될 것"이라고 말했다.
연구진은 암 세포에서만 나타나는 특별한 '염색질 고리' 구조가 유전자 발현 촉진 인자인 '인핸서'와 종양유전자 사이의 상호작용을 형성해 과하게 생기도록 유도하는 '인핸서 납치' 현상에 초점을 뒀다.
특히 게놈간의 공간상 상호작용을 측정할 수 있는 대용량 염색체 구조 포착 'Hi-C' 실험 기법을 활용해 대장암 3차원 게놈 지도를 작성했다. 이를통해 대장암 특이적 3차원 게놈 변화를 환자 개개인별로 분석할 수 있는 AI 기반 알고리즘을 개발했다. 이를 통해 광범위한 규모의 3차원 게놈 구조 변화와 이로 인한 다양한 종양유전자의 활성화를 확인했다.
연구팀은 이를 통해 암 특이적 3차원 게놈 구조의 변화로 인한 종양유전자 활성 현상을 명확히 제시했다. 또 이로 인한 환자 예후와 약물 반응 등 임상적인 특성과의 연관성까지 제시해 맞춤 치료 원천기술을 확보했다.
AI 기반 알고리즘으로 환자 개인 종양 조직으로부터 얻어진 복잡한 신호를 해석해 냈다. 그 결과, 최대 규모인 환자 40명의 종양 조직과 인접한 정상 대장 조직을 사용해 3차원 게놈 지도를 작성했다. 또 한국인 3차원 게놈 지도, 특히 종양 조직에 대한 3차원 게놈 지도를 최초로 해독해냈다.
한편, 연구진은 이번 연구 결과를 국제 학술지 '셀 리포츠(Cell Reports)'에 지난 7월 13일 발표했다.
monarch@fnnews.com 김만기 기자
※ 저작권자 ⓒ 파이낸셜뉴스, 무단전재-재배포 금지